solo per uso di ricerca
P5091 DUB inibitore
N. Cat.S7132
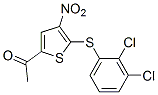
Struttura chimica
Peso molecolare: 348.22
Controllo Qualità
| Target correlati | Proteasome E1 Activating E3 Ligase p97 SUMO E2 conjugating |
|---|---|
| Altro DUB Inibitori | PR-619 IU1 P22077 b-AP15 ML323 LDN-57444 VLX1570 TCID EOAI3402143 ML364 |
Coltura cellulare, trattamento e concentrazione di lavoro
| Linee cellulari | Tipo di saggio | Concentrazione | Tempo di incubazione | Formulazione | Descrizione dellattività | PMID |
|---|---|---|---|---|---|---|
| Sf9 cells | Function assay | Inhibition of recombinant USP7 expressed in Sf9 cells by Ub-CHOP reporter assay, EC50=4.2 μM | 24900381 | |||
| human HCT116 cells | Cytotoxic assay | 72 h | Cytotoxicity against human HCT116 cells after 72 hrs, EC50=11 μM | 24900381 | ||
| multiple myeloma cells | Function assay | Inhibition of USP7 in multiple myeloma cells (unknown origin) by immunohistochemistry, EC50 = 4.2 μM. | 25364867 | |||
| Sf9 | Function assay | 30 mins | Inhibition of full-length recombinant USP7 (unknown origin) expressed in Sf9 cells using Ub-EKL as substrate preincubated for 30 mins followed by substrate addition by fluorescence based assay, IC50 = 4.2 μM. | 28768102 | ||
| Clicca per visualizzare più dati sperimentali sulle linee cellulari | ||||||
Informazioni chimiche, conservazione e stabilità
| Peso molecolare | 348.22 | Formula | C12H7Cl2NO3S2 |
Conservazione (Dalla data di ricezione) | |
|---|---|---|---|---|---|
| N. CAS | 882257-11-6 | Scarica SDF | Conservazione delle soluzioni stock |
|
|
| Sinonimi | P005091 | Smiles | CC(=O)C1=CC(=C(S1)SC2=C(C(=CC=C2)Cl)Cl)[N+](=O)[O-] | ||
Solubilità
|
In vitro |
DMSO
: 28 mg/mL
(80.4 mM)
Water : Insoluble Ethanol : Insoluble |
Calcolatore di Molarità
|
In vivo |
|||||
Calcolatore di formulazione in vivo (Soluzione chiara)
Passo 1: Inserire le informazioni di seguito (Consigliato: Un animale aggiuntivo per tenere conto della perdita durante lesperimento)
Passo 2: Inserire la formulazione in vivo (Questo è solo il calcolatore, non la formulazione. Contattateci prima se non cè una formulazione in vivo nella sezione Solubilità.)
Risultati del calcolo:
Concentrazione di lavoro: mg/ml;
Metodo per preparare il liquido master di DMSO: mg farmaco predissolto in μL DMSO ( Concentrazione del liquido master mg/mL, Vi preghiamo di contattarci prima se la concentrazione supera la solubilità del DMSO del lotto del farmaco. )
Metodo per preparare la formulazione in vivo: Prendere μL DMSO liquido master, quindi aggiungereμL PEG300, mescolare e chiarire, quindi aggiungereμL Tween 80, mescolare e chiarire, quindi aggiungere μL ddH2O, mescolare e chiarire.
Metodo per preparare la formulazione in vivo: Prendere μL DMSO liquido master, quindi aggiungere μL Olio di mais, mescolare e chiarire.
Nota: 1. Si prega di assicurarsi che il liquido sia limpido prima di aggiungere il solvente successivo.
2. Assicurarsi di aggiungere il/i solvente/i in ordine. È necessario assicurarsi che la soluzione ottenuta, nellaggiunta precedente, sia una soluzione limpida prima di procedere allaggiunta del solvente successivo. Metodi fisici come il vortex, gli ultrasuoni o il bagno dacqua calda possono essere utilizzati per facilitare la dissoluzione.
Meccanismo dazione
| Targets/IC50/Ki |
USP7
(Cell-free assay) 4.2 μM(EC50)
USP47
(Cell-free assay) 4.3 μM
|
|---|---|
| In vitro |
P5091 è un tiofene trisostituito con sostituenti diclorofeniltio, nitro e acetilici che mediano l'attività anti-USP7. Questo composto mostra un'attività deubiquitinante potente, specifica e selettiva contro USP7. Al contrario, non inibisce altre DUBs o altre famiglie di cistein proteasi testate (EC50 > 100 mM). Questa sostanza chimica inibisce l'etichettatura di USP7 con HA-UbVME in modo concentrazione-dipendente. La scissione delle catene di poliubiquitina ad alto peso molecolare mediata da USP7 è inibita in modo dose-dipendente da questo composto. Inoltre, inibisce la scissione delle catene di ubiquitina poli-K48-legate mediata da USP7, ma non da USP2 o USP8. L'inibizione di USP7 da parte di questo composto induce la poliubiquitinazione di HDM2 e accelera la degradazione di HDM2. Inibisce l'attività deubiquitinante di USP7, senza bloccare l'attività del proteasoma nelle cellule MM. Questa sostanza chimica induce una diminuzione dose-dipendente della vitalità di varie linee cellulari MM. Supera la crescita delle cellule MM indotta dalle cellule stromali del midollo osseo. Questo composto ha diminuito i livelli di HDM2 e HDMX, oltre ad aver aumentato i livelli di p53 e p21. Nel complesso, la citotossicità indotta da questo composto è mediata in parte dall'asse di segnalazione HDM2-p21 e, sebbene p53 sia sovraregolato in risposta a questo trattamento chimico, l'attività citotossica di esso non dipende da p53. |
| Saggio chinasico |
Saggi di Ubiquitin Proteasi
|
|
Enzimi ricombinanti in 20 mM Tris-HCl (pH 8.0), 2 mM CaCl2 e 2 mM β-mercaptoetanolo vengono incubati con intervalli di dosi di P5091 per 30 min in una piastra a 96 pozzetti prima dell'aggiunta di Ub-PLA2 e NBD C6-HPC o Ub-EKL e substrato EKL. La liberazione di un prodotto fluorescente entro il range lineare del saggio viene monitorata utilizzando un lettore di piastre a fluorescenza Perkin Elmer Envision. Il veicolo (2% [v/v] DMSO) e 10 mM di N-etilmaleimide (NEM) sono inclusi come controlli.
|
|
| In vivo |
Negli studi su modelli animali di tumore, P5091 è ben tollerato, inibisce la crescita tumorale e prolunga la sopravvivenza. La combinazione di questo composto con l'inibitore HDAC SAHA innesca un'attività anti-MM sinergica. |
Riferimenti |
|
Applicazioni
| Metodi | Biomarcatori | Immagini | PMID |
|---|---|---|---|
| Western blot | USP7 / p53 / p21 HAUSP / N-Myc |
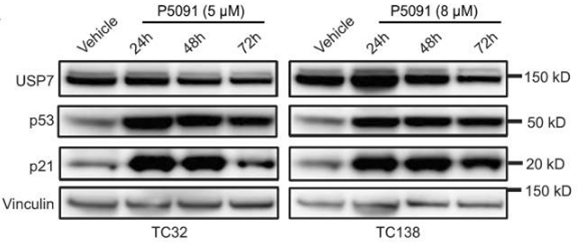
|
30045945 |
| Immunofluorescence | PTEN |
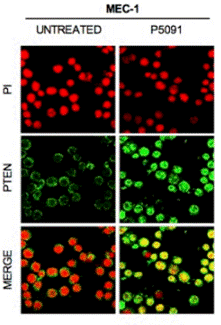
|
28418900 |
| Growth inhibition assay | Cell viability |
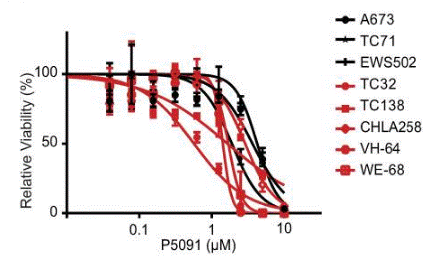
|
30045945 |
Supporto tecnico
Istruzioni per la manipolazione
Tel: +1-832-582-8158 Ext:3
Per qualsiasi altra domanda, si prega di lasciare un messaggio.
