solo per uso di ricerca
T0901317 Liver X Receptor agonista
N. Cat.S7076

Struttura chimica
Peso molecolare: 481.33
Controllo Qualità
| Target correlati | Dehydrogenase HSP Transferase P450 (e.g. CYP17) PDE phosphatase PPAR Vitamin Carbohydrate Metabolism Mitochondrial Metabolism |
|---|---|
| Altro Liver X Receptor Inibitori | GW3965 Hydrochloride LXR-623 (WAY-252623) GSK2033 SR9243 Abequolixron (RGX-104) AZ876 GSK3987 Desmosterol |
Coltura cellulare, trattamento e concentrazione di lavoro
| Linee cellulari | Tipo di saggio | Concentrazione | Tempo di incubazione | Formulazione | Descrizione dellattività | PMID |
|---|---|---|---|---|---|---|
| THP1 cells | Function assay | Stimulation of [3H]cholesterol efflux in human THP1 foam cells loaded with ac-LDL, EC50=3 nM | ||||
| COS7 cells | Function assay | 16 h | Agonist activity at human LXRbeta receptor transfected in COS7 cells after 16 hrs by reporter transactivation assay, EC50=11 nM | |||
| CHO cells | Function assay | Agonist activity at human LXR beta receptor expressed in CHO cells by reporter assay, EC50=16 nM | ||||
| HEK293 cells | Function assay | Agonist activity at human LXRbeta in HEK293 cells assessed as Gal4 transactivation, EC50=19 nM | ||||
| mouse J774 cells | Function assay | Agonist activity at LXRbeta in mouse J774 cells assessed as upregulation of ABCA1 mRNA expression, EC50=0.027 μM | ||||
| mouse J774A1 cells | Function assay | 18 h | Induction of ABCA1 mRNA expression in mouse J774A1 cells after 18 hrs by RT-PCR, EC50=0,034 μM | |||
| HuH7 cells | Function assay | Upregulation of SREB1c mRNA level in human HuH7 cells, EC50=0.036 μM | ||||
| HepG2 cells | Function assay | Effect on SREBP1c gene expression in human HepG2 cells, EC50=0.061 μM | ||||
| SH-SY5Y cells | Function assay | 24 h | Agonist activity at human LXRbeta expressed in human SH-SY5Y cells co-transfected with Gal4-LBD after 24 hrs by luciferase reporter gene assay, EC50=0.075 μM | |||
| CV1 cells | Function assay | Transactivation of LXRbeta (unknown origin) expressed in CV1 cells by luciferase reporter gene assay, EC50=0.14 μM | ||||
| human H4 cells | Function assay | Effect on gamma-secretase activity in human H4 cells expressing APP695 assessed as increase in Amyloid beta-42 formation LPECL assay, EC50=3.6 μM | ||||
| Clicca per visualizzare più dati sperimentali sulle linee cellulari | ||||||
Informazioni chimiche, conservazione e stabilità
| Peso molecolare | 481.33 | Formula | C17H12F9NO3S |
Conservazione (Dalla data di ricezione) | |
|---|---|---|---|---|---|
| N. CAS | 293754-55-9 | Scarica SDF | Conservazione delle soluzioni stock |
|
|
| Sinonimi | N/A | Smiles | C1=CC=C(C=C1)S(=O)(=O)N(CC(F)(F)F)C2=CC=C(C=C2)C(C(F)(F)F)(C(F)(F)F)O | ||
Solubilità
|
In vitro |
DMSO
: 96 mg/mL
(199.44 mM)
Ethanol : 96 mg/mL Water : Insoluble |
Calcolatore di Molarità
|
In vivo |
|||||
Calcolatore di formulazione in vivo (Soluzione chiara)
Passo 1: Inserire le informazioni di seguito (Consigliato: Un animale aggiuntivo per tenere conto della perdita durante lesperimento)
Passo 2: Inserire la formulazione in vivo (Questo è solo il calcolatore, non la formulazione. Contattateci prima se non cè una formulazione in vivo nella sezione Solubilità.)
Risultati del calcolo:
Concentrazione di lavoro: mg/ml;
Metodo per preparare il liquido master di DMSO: mg farmaco predissolto in μL DMSO ( Concentrazione del liquido master mg/mL, Vi preghiamo di contattarci prima se la concentrazione supera la solubilità del DMSO del lotto del farmaco. )
Metodo per preparare la formulazione in vivo: Prendere μL DMSO liquido master, quindi aggiungereμL PEG300, mescolare e chiarire, quindi aggiungereμL Tween 80, mescolare e chiarire, quindi aggiungere μL ddH2O, mescolare e chiarire.
Metodo per preparare la formulazione in vivo: Prendere μL DMSO liquido master, quindi aggiungere μL Olio di mais, mescolare e chiarire.
Nota: 1. Si prega di assicurarsi che il liquido sia limpido prima di aggiungere il solvente successivo.
2. Assicurarsi di aggiungere il/i solvente/i in ordine. È necessario assicurarsi che la soluzione ottenuta, nellaggiunta precedente, sia una soluzione limpida prima di procedere allaggiunta del solvente successivo. Metodi fisici come il vortex, gli ultrasuoni o il bagno dacqua calda possono essere utilizzati per facilitare la dissoluzione.
Meccanismo dazione
| Targets/IC50/Ki |
LXR
(Cell reporter assay) 50 nM(EC50)
FXR
5 μM(EC50)
|
|---|---|
| In vitro |
T0901317 agisce tramite LXR e, in concerto con il suo partner di eterodimerizzazione RXR, induce l'espressione del trasportatore inverso del colesterolo ABCA1. Questo composto sovraregola l'espressione del gene ABCA1 associato alla regolazione dell'efflusso di colesterolo e al Metabolismo delle HDL. Mostra un EC50 di ~ 5 M per l'attivazione dei recettori X farnesoidi degli acidi biliari (FXRs), 10 volte pi potente dell'acido chenodeossicolico, ligando naturale di FXR. Questa sostanza chimica anche un ligando ad alta affinit per il recettore xenobiotico pregnane X receptor (PXR). Si lega e attiva PXR con la stessa potenza nanomolare con cui stimola l'attivit LXR. Questo composto induce l'espressione non solo dei geni bersaglio LXR, ma anche dei geni bersaglio PXR in cellule e animali, incluso il recettore spazzino CD36. Diminuisce la produzione di amiloide- nei neuroni primari in vitro. Si scoperto che questa sostanza chimica si lega direttamente a ROR e ROR con alta affinit (Ki = 132 e 51 nM, rispettivamente), con conseguente modulazione della capacit del recettore di interagire con le proteine cofattore trascrizionali. Reprime la transattivazione dipendente da ROR / dei geni reporter responsivi a ROR e nelle cellule HepG2 riduce il reclutamento del coattivatore del recettore steroideo-2 da parte di ROR a un gene bersaglio ROR endogeno (G6Pase). |
| In vivo |
Il trattamento con T0901317 di topi APP23 di 11 settimane per 6 giorni mostra un aumento significativo dell'espressione di ABCA1 e una diminuzione del rapporto tra i prodotti di clivaggio di APP (sAPP) - e sAPP - solubile. Ancora pi importante, questo composto causa una riduzione statisticamente significativa dei livelli di A 40 solubile e di A 42 nel cervello di questi topi. |
Riferimenti |
|
Applicazioni
| Metodi | Biomarcatori | Immagini | PMID |
|---|---|---|---|
| Western blot | LXR-β / LXR-α EGFR / p-PI3K p55 / p-PI3K p85 / PI3K p85 / p-PDK1 / PDK1 |
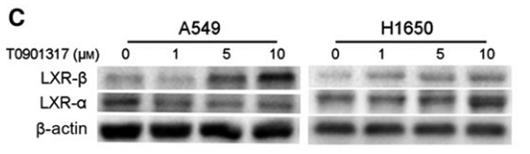
|
28097086 |
Supporto tecnico
Istruzioni per la manipolazione
Tel: +1-832-582-8158 Ext:3
Per qualsiasi altra domanda, si prega di lasciare un messaggio.
