réservé à la recherche
YM201636 PIKfyve inhibiteur
N° Cat.S1219
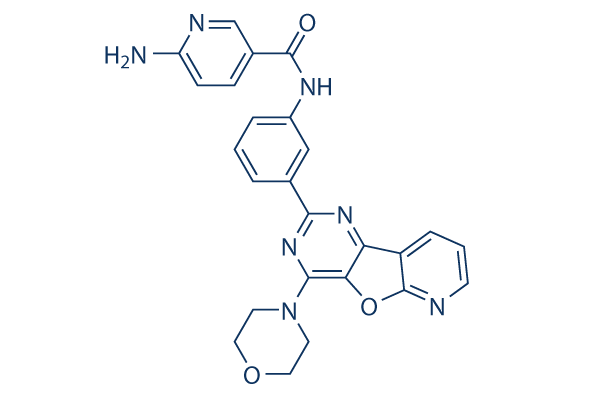
Structure chimique
Poids moléculaire: 467.48
Contrôle qualité
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| SJ-GBM2 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SJ-GBM2 cells | 29435139 | |||
| A673 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for A673 cells | 29435139 | |||
| SK-N-MC | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SK-N-MC cells | 29435139 | |||
| BT-37 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for BT-37 cells | 29435139 | |||
| NB-EBc1 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for NB-EBc1 cells | 29435139 | |||
| U-2 OS | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for U-2 OS cells | 29435139 | |||
| Saos-2 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Saos-2 cells | 29435139 | |||
| NB1643 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for NB1643 cells | 29435139 | |||
| LAN-5 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for LAN-5 cells | 29435139 | |||
| Rh18 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Rh18 cells | 29435139 | |||
| OHS-50 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for OHS-50 cells | 29435139 | |||
| RD | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for RD cells | 29435139 | |||
| VERO-E6 | Toxicity assay | 48 hrs | Toxicity CC50 against VERO-E6 cells determined at 48 hours by high content imaging (same conditions as 2_LEY without exposure to 0.01 MOI SARS CoV-2 virus), CC50=0.01μM | ChEMBL | ||
| VERO-E6 | Function assay | 48 hrs | Determination of IC50 values for inhibition of SARS-CoV-2 induced cytotoxicity of VERO-E6 cells after 48 hours exposure to 0.01 MOI SARS CoV-2 virus by high content imaging, IC50=4.73μM | ChEMBL | ||
| Cliquez pour voir plus de données expérimentales sur les lignées cellulaires | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 467.48 | Formule | C25H21N7O3 |
Stockage (À partir de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 371942-69-7 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | N/A | Smiles | C1COCCN1C2=NC(=NC3=C2OC4=C3C=CC=N4)C5=CC(=CC=C5)NC(=O)C6=CN=C(C=C6)N | ||
Solubilité
|
In vitro |
DMSO
: 12 mg/mL
(25.66 mM)
Water : Insoluble Ethanol : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Entrez les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Entrez la formulation in vivo (Ceci nest que le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuiteμL PEG300, mélanger et clarifier, ajouter ensuiteμL Tween 80, mélanger et clarifier, ajouter ensuite μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuite μL Huile de maïs, mélanger et clarifier.
Remarque : 1. Assurez-vous que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue lors de lajout précédent est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
PIKfyve
(Cell-free assay) 33 nM
p110α
(Cell-free assay) 3.3 μM
|
|---|---|
| In vitro |
YM201636 inhibe puissamment la PIKfyve de mammifères avec une IC50 de 33 nM mais pas l'orthologue de levure Fab1 avec une IC50 >5 μM, montrant une sélectivité environ 100 fois supérieure pour PtdIns3P p110α avec une IC50 de 3 μM. Ce composé (0,8 μM) diminue significativement la production de PtdIns(3,5)P2 de 80% dans les cellules NIH3T3 privées de sérum, suivies d'une stimulation sérique sans effet sur la phosphorylation de la protéine kinase B (PKB) Ser 473 stimulée par le sérum. Il altère réversiblement le trafic endosomal dans les cellules NIH3T3 en bloquant PIKfyve et la production de PtdIns(3,5)P2, mimant l'effet produit par l'épuisement de PIKfyve avec le siRNA. Ce produit chimique (0,8 μM) réduit également significativement le bourgeonnement des rétrovirus des cellules de 80%, apparemment en interférant avec le complexe de tri endosomal requis pour le transport (ESCRT). Dans les adipocytes 3T3L1, il inhibe l'absorption basale et stimulée par l'insuline du 2-désoxyglucose avec une IC50 de 54 nM, avec une inhibition presque complète à des doses aussi faibles que 160 nM. Ce composé (0,1 μM) a également été montré pour bloquer complètement l'activation dépendante de l'insuline de la PI 3-kinase de classe IA. Bien que non impliqué dans la prolifération et la migration dépendantes de NPM-ALK, il (0,4 μM) réduit fortement les capacités invasives des cellules exprimant NPM-ALK et leur capacité à dégrader la matrice extracellulaire. Le traitement avec ce produit chimique bloque le recyclage continu des protéines jonctionnelles claudine-1 et claudine-2 dans les cellules MDCK, entraînant l'accumulation intracellulaire et le retard de la formation de la barrière épithéliale.
|
Références |
|
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
